Importar archivo .LAS con lasio, analizarlos y graficarlos en formato log

En este artículo vamos a describir cómo cargar un archivo .las, (archivo estándar de la industria del petróleo y gas), con lasio, convertirlo en pandas dataframe, hacer el control de calidad de los datos, y graficar las curvas con matplotlib.
Bienvenidas/os!!, muchas gracias por visitarnos, si están comenzando en Python y este es el primer artículo al que entran en nuestro sitio, les recomendamos visitar Primeros Pasos.
ℹ️ Recuerden que todos los ejemplos de códigos en nuestro sitio web van a encontrarlos en nuestro repositorio GitHub. Los códigos de este artículo los encontrarán en LAS_lasio_pandas_matplotlib.ipynb.
ℹ️ Toda la información de archivos .las/.csv contenida en este sitio web es información liberada por sus propietarios.En la industria del petróleo y gas, la información de los registros de pozos es almacenada generalmente en archivos .LAS, LIS/TAP, ó DLIS/DLS. En este artículo, veremos como importar y trabajar con archivos .las utilizando la librería lasio.
CARGA DE LOS DATOS
La carga/importación la haremos utilizando la librería lasio.
Importamos TODAS las librerías necesarias para cargar, manejar y visualizar los datos:
ℹ️ Es aconsejable importar todas las librerías que vayamos necesitando a lo largo de todo el notebook en una misma celda al comienzo del mismo, y no tener líneas con import en varias celdas.ℹ️ En este caso utilizamos mplcursors, que es una librería que trabaja sobre matplotlib, y que nos permite ver los valores del gráfico al movernos sobre él. En el caso de no tenerla, pueden instalarla tipeando pip install mplcursors.
import numpy as np
import pandas as pd
import lasio
import matplotlib.pyplot as plt
from matplotlib.ticker import StrMethodFormatter
from matplotlib.ticker import MaxNLocator
%matplotlib inline
import mplcursors
%matplotlib notebook
Ahora vamos a importar un archivo las y lo asignamos a la variable las
# Importar archivo LAS
las = lasio.read(r'D:\Pablo\Python\working_files\Equinor\15_9-23.las')
Veamos qué curvas tiene el archivo tipeando:
# VER INFORMACION DE LAS CURVAS EN EL ARCHIVO .las (opción 1)
print(las.curves)
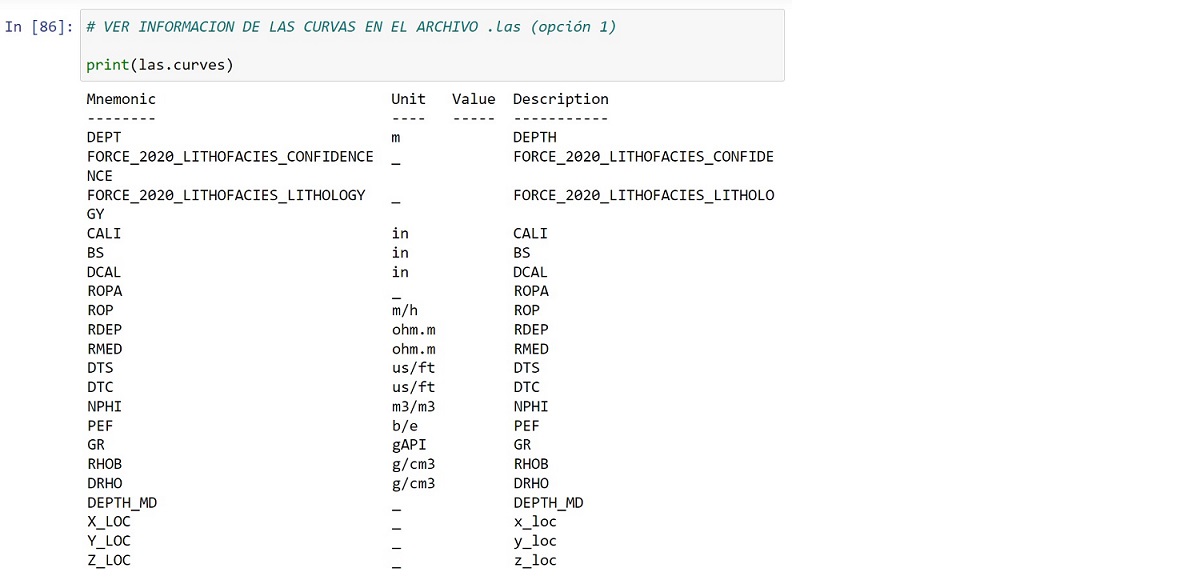
También podemos ver datos del pozo tipeando:
for item in las.well:
print(f'{item.descr} ({item.mnemonic}): {item.value}')
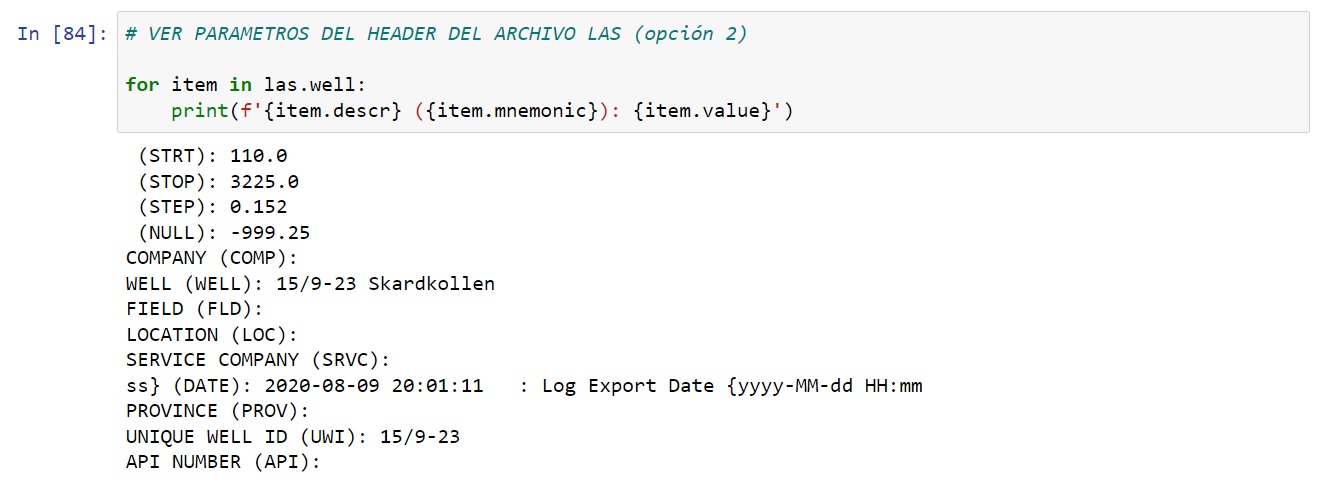
CONTROL DE CALIDAD DE LOS DATOS
Una vez que pegamos una mirada a los datos del pozo, vamos a convertir el objeto LASIO a pandas dataframe para trabajar con los valores de las curvas. lasio tiene el método df() para hacerlo de manera sencilla:
well = las.df()
well = well.reindex(sorted(well.columns), axis=1) # Ordenamos las columnas alfabéticamente
pd.set_option('display.max_columns', 200) # Muestra las primeras 200 columnas
well # tambien podemos utilizar well.head() ó well.tail()
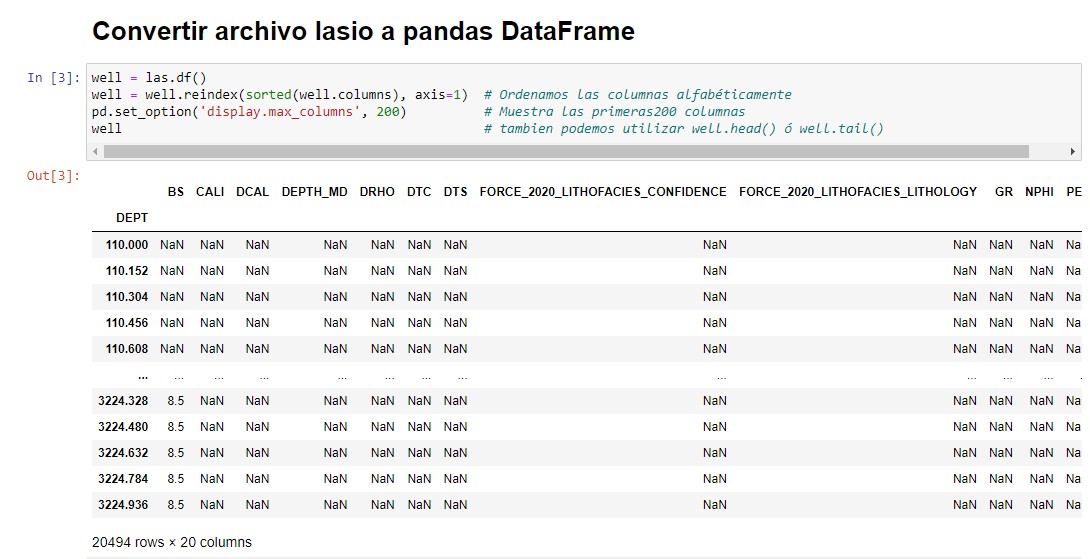
Ahora podemos trabajar con pandas. (no es objetivo de este artículo cubrir esta parte, si quieren ver ejemplos de código para hacer control de calidad en pandas, por favor visiten nuestro artículo Manejo de datos utilizando pandas).
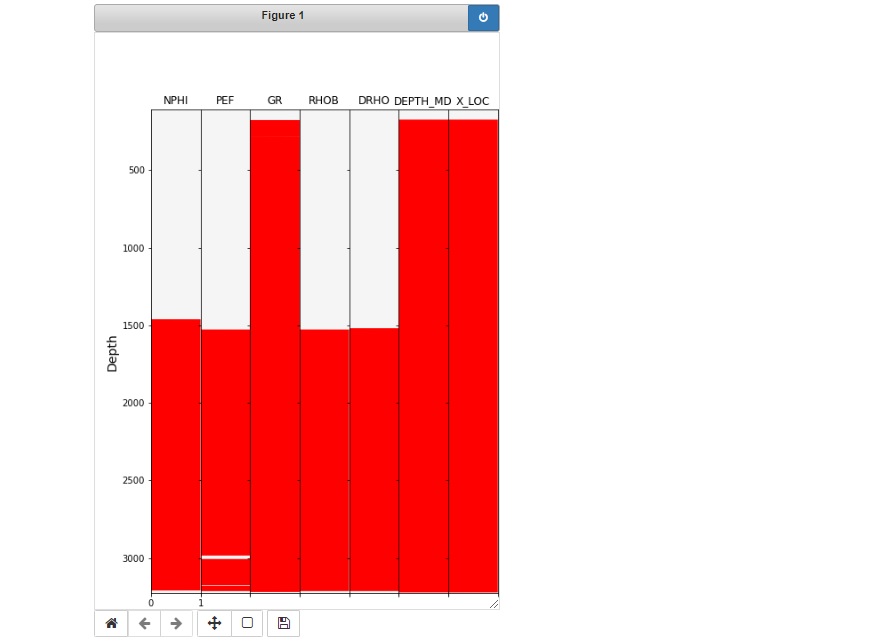
Lo siguiente es lo que todas/os las/os petrofísicas/os o analistas de perfiles estaban esperando, visualizar un gráfico a modo de log con algunas curvas para tratar de ver si tiene y/o dónde está el tramo principal del pozo. (El gráfico que elegimos va a pintar de rojo los tramos con valores, y pintará en gris los que no tienen, o mejor dicho, tienen valores nulos).
Este gráfico lo tomamos de aquí, https://andymcdonaldgeo.medium.com/loading-and-displaying-well-log-data-b9568efd1d8, y lo modificamos ya que en el original alguna de las curvas del tramo principal ó TP, debía coincidir con las primeras 7 columnas del dataframe, sino podíamos llegar a malinterpretar las profundidades del TP.
Si bien el plot símil perfiles combinados es más conveniente, en nuestra opinión, para visualizar el TP este gráfico es interesante porque muestra los valores nulos y puede llegar a mostrar pequeños gaps.
# Con este codigo vamos a plotear 7 curvas para poder encontrar el TP (tramo principal) del log
# Como vamos a plotearlas según su orden en el archivo lasio (INDEXING), vamos a utilizar el INDEXING para elegirlas.
# Con la variable primercol vamos a definir la primer curva utilizando el INDEXING.
well_nan = well.notnull() * 1
primercol = 12 # Primer curva que va a plotear
fig = plt.subplots(figsize=(7,10))#Set up the plot axes
ax1 = plt.subplot2grid((1,7), (0,0), rowspan=1, colspan = 1)
ax2 = plt.subplot2grid((1,7), (0,1), rowspan=1, colspan = 1)
ax3 = plt.subplot2grid((1,7), (0,2), rowspan=1, colspan = 1)
ax4 = plt.subplot2grid((1,7), (0,3), rowspan=1, colspan = 1)
ax5 = plt.subplot2grid((1,7), (0,4), rowspan=1, colspan = 1)
ax6 = plt.subplot2grid((1,7), (0,5), rowspan=1, colspan = 1)
ax7 = plt.subplot2grid((1,7), (0,6), rowspan=1, colspan = 1)
columns = well_nan.columns
axes = [ax1, ax2, ax3, ax4, ax5, ax6, ax7]
for i, ax in enumerate(axes):
ax.plot(well_nan.iloc[:,i], well_nan.index, lw=0)
ax.set_ylim(las.well.STOP.value, las.well.STRT.value)
ax.set_xlim(0, 1)
ax.set_title(columns[i+primercol-1]) # i+18
ax.set_facecolor('whitesmoke')
ax.fill_betweenx(well_nan.index, 0, well_nan.iloc[:,i+primercol-1], facecolor='red') # i+18
# Remove tick labels from each subplot
if i > 0:
plt.setp(ax.get_yticklabels(), visible = False)
plt.setp(ax.get_xticklabels(), visible = False)
ax1.set_ylabel('Depth', fontsize=14)
plt.subplots_adjust(wspace=0)
plt.show()
Antes de plotear los perfiles combinados, vamos a declarar las variables a plotear. Esto lo hacemos por 2 motivos:
- Los nombres de las curvas son diferentes según la compañía de perfilaje.
- En el caso de que no contemos con una de las curvas incluidas en el plot, esta celda va a generarla con valores nulos (esto es porque si matplotlib no encuentra ninguna curva, va a dar error y no va a plotear nada)
# DECLARACION DE VARIABLES PARA PLOTEAR PERFILES COMBINADOS
bitsize = 8.75
## ax1
crv_cal = 'CALI' # CALIPER
crv_bit = 'BS' # BIT SIZE
crv_gr = 'GR' # GAMMA RAY
crv_sp = 'SP' # SPONTANEOUS POTENTIAL
# ax2
crv_dp_res = 'RDEP' # DEEP RESISTIVITY
crv_sh_res = 'RMED' # SHALLOW RESISTIVITY
#ax3
crv_neu_por = 'NPHI' # NEUTRON POROSITY
crv_den_bulk = 'RHOB' # BULK DENSITY
##############################################################
if crv_cal in well.columns:
pass
else:
well[crv_cal]=np.nan
##############
if crv_bit in well.columns:
pass
else:
well[crv_bit]=bitsize
##############
if crv_gr in well.columns:
pass
else:
well[crv_gr]=np.nan
##############
if crv_sp in well.columns:
pass
else:
well[crv_sp]=np.nan
##############
if crv_dp_res in well.columns:
pass
else:
well[crv_dp_res]=np.nan
##############
if crv_sh_res in well.columns:
pass
else:
well[crv_sh_res]=np.nan
##############
if crv_neu_por in well.columns:
pass
else:
well[crv_neu_por]=np.nan
##############
if crv_den_bulk in well.columns:
pass
else:
well[crv_den_bulk]=np.nan
##############
GRAFICAR PLOT PERFILES COMBINADOS
En la celda de abajo está el código para plotear 3 tracks:
- Curvas convencionales(GR,CAL,BIT,SP)
- Resistividades(RDEP,RMED)
- Porosidades(BULKDEN,PORNEU)
Además, es aconsejable tener en cuenta lo siguiente:
- Para cambiar TOP y BOT del plot, modificar los valores top_plot y bot_plot.
- La cantidad de labels de cada track se modifica con las variables ax1_ticks,ax2_ticks,etc.
- Los valores máximos y mínimos de las curvas representan en realidad su ubicación en el track (mínimo=izquierda, máximo=derecha), por ejemplo los límites gr_min=0, gr_max=200 serían para la curva GR, pero para la curva Porosidad Neutron serían neupor_min = .6, neupor_max = 0.
top_plot = las.well.STRT.value # Valor default TOP del plot (modificar si es necesario)
bot_plot = las.well.STOP.value # Valor default BOTTOM del plot (modificar si es necesario)
ax1_ticks = 6 # Track Convencionales
ax2_ticks = 6 # Track Resistividades
ax3_ticks = 7 # Track Porosidades
fig, ax = plt.subplots(figsize=(25,25))#Set up the plot axes
ax1 = plt.subplot2grid((1,6), (0,0), rowspan=1, colspan = 1)
ax11 = ax1.twiny()
ax12 = ax1.twiny()
ax2 = plt.subplot2grid((1,6), (0,1), rowspan=1, colspan = 1, sharey = ax1)
ax21 = ax2.twiny()
ax3 = plt.subplot2grid((1,6), (0,2), rowspan=1, colspan = 1, sharey = ax1) #
ax31 = ax3.twiny()
################################################################################
### CAL ###
cal_min = 10
cal_max = 20
ax1.plot(well[crv_cal], well.index, color = "black", linewidth = 0.5)
ax1.set_ylabel("Depth (m)")
if abs(top_plot-bot_plot) > 5000:
ax1.yaxis.set_major_locator(MaxNLocator(round(abs(top_plot-bot_plot)/999)))
elif abs(top_plot-bot_plot) > 2000 and abs(top_plot-bot_plot) <= 5000:
ax1.yaxis.set_major_locator(MaxNLocator(round(abs(top_plot-bot_plot)/450)))
elif abs(top_plot-bot_plot) > 500 and abs(top_plot-bot_plot) <= 2000:
ax1.yaxis.set_major_locator(MaxNLocator(round(abs(top_plot-bot_plot)/99)))
elif abs(top_plot-bot_plot) <= 500 and abs(top_plot-bot_plot) > 200:
ax1.yaxis.set_major_locator(MaxNLocator(round(abs(top_plot-bot_plot)/49)))
else:
ax1.yaxis.set_major_locator(MaxNLocator(round(abs(top_plot-bot_plot)/24)))
ax1.set_xlabel(f"Caliper {well[crv_cal].name}")
ax1.set_xlim(cal_min, cal_max)
ax1.xaxis.label.set_color("black")
ax1.tick_params(axis='x', colors="black")
ax1.spines["top"].set_edgecolor("black")
ax1.set_xticks(np.arange(cal_min, cal_max+1, ((cal_max-cal_min)/(ax1_ticks-1))))
ax1.xaxis.set_major_formatter(StrMethodFormatter('{x:.0f}'))
ax1.fill_betweenx(well.index, well[crv_bit], well[crv_cal], where=well[crv_bit]>=well[crv_cal], interpolate=True, color='brown')
ax1.fill_betweenx(well.index, well[crv_bit], well[crv_cal], where=well[crv_bit]<=well[crv_cal], interpolate=True, color='#a6ecfa')
################################################################################
### GR ###
gr_min=0
gr_max=200
ax11.plot(well[crv_gr], well.index, color = "green", linewidth = 1.5)
ax11.set_xlabel(f"Gamma Ray ({well[crv_gr].name})")
ax11.xaxis.label.set_color("green")
ax11.set_xlim(gr_min, gr_max)
ax11.tick_params(axis='x', colors="green")
ax11.spines["top"].set_position(("axes", 1.12))
ax11.spines["top"].set_visible(True)
ax11.spines["top"].set_edgecolor("green")
ax11.title.set_color('green')
ax11.set_xticks(np.arange(gr_min, gr_max+1, ((gr_max-gr_min)/(ax1_ticks-1))))
ax11.xaxis.set_major_formatter(StrMethodFormatter('{x:.0f}'))
################################################################################
### SP ###
sp_min=-80
sp_max=20
ax12.plot(well[crv_sp], well.index, color = "red", linewidth = 1.5)
ax12.set_xlabel(f"SP ({well[crv_sp].name})")
ax12.xaxis.label.set_color("red")
ax12.set_xlim(sp_min,sp_max)
ax12.tick_params(axis='x', colors="r")
ax12.spines["top"].set_position(("axes", 1.07))
ax12.spines["top"].set_visible(True)
ax12.spines["top"].set_edgecolor("r")
ax12.title.set_color('r')
ax12.set_xticks(np.arange(sp_min, sp_max+1, ((sp_max-sp_min)/(ax1_ticks-1))))
ax12.xaxis.set_major_formatter(StrMethodFormatter('{x:.0f}'))
################################################################################
### RESD ###
res_min=0.1
res_max=100
ax2.plot(well[crv_dp_res], well.index, color = "blue", linewidth = 2)
ax2.set_xlabel(f"Deep Resistivity ({well[crv_dp_res].name})")
ax2.set_xlim(res_min, res_max)
ax2.xaxis.label.set_color("blue")
ax2.tick_params(axis='x', colors="blue")
ax2.spines["top"].set_edgecolor("blue")
ax2.set_xticks(np.arange(res_min, res_max+.1, (res_max/(ax2_ticks-1))))
ax2.set_xscale("log")
ax2.grid(True, which="both", axis='x')
ax2.xaxis.set_major_formatter(StrMethodFormatter('{x:.1f}'))
################################################################################
### RESS ###
ax21.plot(well[crv_sh_res], well.index, color = "r", linewidth = 0.5)
ax21.set_xlabel(f"Shallow Resistivity ({well[crv_sh_res].name})")
ax21.set_xlim(res_min, res_max)
ax21.xaxis.label.set_color("r")
ax21.spines["top"].set_position(("axes", 1.07))2.6952.+6
ax21.spines["top"].set_visible(True)
ax21.tick_params(axis='x', colors="r")
ax21.spines["top"].set_edgecolor("r")
ax21.set_xticks(np.arange(res_min, res_max+.1, ((res_max-res_min)/(ax2_ticks-1))))
ax21.set_xscale("log")
ax21.xaxis.set_major_formatter(StrMethodFormatter('{x:.1f}'))
################################################################################
### DEN BULK ###
denbulk_min = 1.65
denbulk_max = 2.65
ax3.plot(well[crv_den_bulk], well.index, color = "red", linewidth = 1.5)
ax3.set_xlabel(f"Density Bulk ({well[crv_den_bulk].name})")
ax3.set_xlim(denbulk_min,denbulk_max)
ax3.xaxis.label.set_color("red")
ax3.tick_params(axis='x', colors="r")
ax3.spines["top"].set_edgecolor("r")
ax3.set_xticks(np.arange(denbulk_max, denbulk_min+.01, ((denbulk_min-denbulk_max)/(ax3_ticks-1))))
ax3.xaxis.set_major_formatter(StrMethodFormatter('{x:.2f}'))
################################################################################
### NEU POR ###
neupor_min = .6
neupor_max = 0
ax31.plot(well[crv_neu_por], well.index, color = "blue", linewidth = 1.5)
ax31.set_xlabel(f"Neutron Porosity ({well[crv_neu_por].name})")
ax31.xaxis.label.set_color("b")
ax31.set_xlim(neupor_min,neupor_max)
ax31.tick_params(axis='x', colors="blue")
ax31.spines["top"].set_position(("axes", 1.07))
ax31.spines["top"].set_visible(True)
ax31.spines["top"].set_edgecolor("blue")
ax31.set_xticks(np.arange(neupor_max, neupor_min+.01, (abs(neupor_max-neupor_min)/(ax3_ticks-1))))
ax31.xaxis.set_major_formatter(StrMethodFormatter('{x:.2f}'))
################################################################################
fig.suptitle('\nPERFILES COMBINADOS\n\n'+"POZO: "+las.well.WELL.value+"\n"+"YACIMIENTO: "+las.well.FLD.value+"\n"+"COMPAÑIA: "+las.well.COMP.value,size=26,
x=0.27,y=1)
# Common functions for setting up the plot can be extracted into
# a for loop. This saves repeating code.
for ax in [ax1, ax2, ax3]:
ax.set_ylim(bot_plot, top_plot)
ax.grid(which='major', color='lightgrey', linestyle='-')
ax.xaxis.set_ticks_position("top")
ax.xaxis.set_label_position("top")
ax.spines["top"].set_position(("axes", 1.02))
for ax in [ax2, ax3]:
plt.setp(ax.get_yticklabels(), visible = False)
fig.subplots_adjust(wspace = 0.2)
mplcursors.cursor(hover=True)
plt.tight_layout()
plt.show()
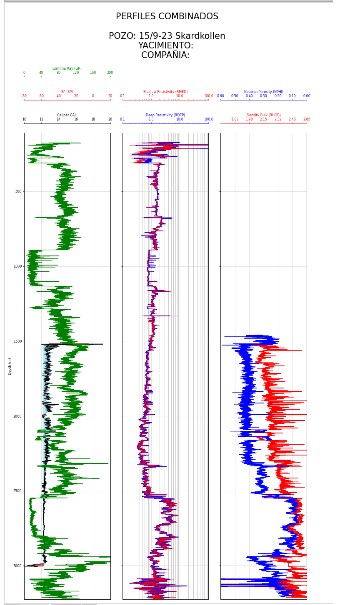
Resumiendo, hemos importado los datos de un archivo .LAS utilizando lasio, para luego insertarlos dentro de un dataframe pandas con el cual hicimos el control de calidad de la información de las curvas. Luego hemos hecho un primer plot para distinguir el tramo principal. Seguidamente realizamos una conveniente declaración de variables y luego, para finalizar, ploteamos un gráfico incluyendo los perfiles combinados.VOLVER⤴️
Les agradecemos su tiempo y esperamos fervientemente que hayan disfrutado este artículo. Si tienen alguna consulta, desean hacer algún comentario o sugerencia para mejorar el contenido, o simplemente indicarles qué les pareció este artículo, debajo pueden hacerlo.
Esperamos reencontrarlos en algún otro artículo del sitio. Hasta luego!
Tags
Recent Posts