Emulación de curvas de pozo utilizando RNA - Entrenamiento y Aplicación del modelo con Keras - Parte 3
Uno de los usos más comunes que le dan los petrofísicos y analistas de perfiles a las redes neuronales, según nuestro entender, es la emulación de datos de interés para un pozo de petróleo o gas, a partir del entrenamiento de redes neuronales con datos registrados en ese mismo pozo, y/o en otros pozos cercanos donde las litologías atravesadas en todos ellos sean análogas (mismas formaciones, espesores similares, etc).
Bienvenidas/os!!, muchas gracias por visitarnos, si están comenzando en Python y este es el primer artículo al que entran en nuestro sitio, les recomendamos visitar Primeros Pasos.
ℹ️ Recuerden que todos los ejemplos de códigos en nuestro sitio web van a encontrarlos en nuestro repositorio GitHub. Los códigos de este artículo los encontrarán en ANN_supervised_regression_training.ipynb.
POZO ENTRENADOR
ENTRENAMIENTO Y EVALUACION DEL MODELO CON KERAS
Llegamos a la parte final de nuestra tarea, el entrenamiento del modelo. Aquí elegiremos nuestra curva target, y las curvas que utilizaremos como features. También, definiremos las profundidades donde realizaremos el entrenamiento. Luego, realizaremos dicho entrenamiento del modelo con Keras. Para finalizar, tomaremos el modelo y crearemos una curva emulada, la cual será comparada con la curva target en un gráfico tipo plot.
INDICE
- Creacion de los df con las curvas que utilizaremos como features y la curva target, ambas en el tramo del pozo conveniente
- Creación de funciones para graficar el modelo
- Keras: Entrenamiento del modelo (fit)
- Keras: Aplicación del modelo - Obtención de la curva emulada (predict)
- Plot para comparar curva target vs. curva emulada
Creacion de los df con las curvas que utilizaremos como features y la curva target, ambas en el tramo del pozo conveniente
Antes de entrenar el modelo, necesitamos definir la/s curva/s que queremos emular, o target, y las curvas que utilizaremos como input para tal fin, o features.
En la siguiente celda, vamos a declarar cual será la curva target, y cuales las curvas features (hasta 10). Además, definiremos el tramo del pozo donde vamos a realizar el entrenamiento.
# DECLARACION DE CURVAS FEATURES y TARGET
##############################################################
# TRAMO A PROCESAR
well_top = 1140
well_bot = 2990
#######################################
# CURVAS PARA ENTRENAMIENTO CON KERAS
crv_target = 'NPHI'
crv_feature1 = 'CALI'
crv_feature2 = 'GR'
crv_feature3 = 'DTC'
crv_feature4 = 'PEF'
crv_feature5 = 'RHOB'
crv_feature6 = 'RDEP'
crv_feature7 = 'RMED'
crv_feature8 = 'SP'
crv_feature9 = 'NADA'
crv_feature10 = 'NADA'
##############################################################
well_train = well[(well.DEPTH >= well_top) & (well.DEPTH <= well_bot)]
well_train['target'] = well_train[crv_target]
if 'features_list' in locals():
del features_list
else:
features_list = []
features_list = []
###
if crv_feature1 in well_train.columns:
features_list.append(crv_feature1)
else:
pass
if crv_feature2 in well_train.columns:
features_list.append(crv_feature2)
else:
pass
if crv_feature3 in well_train.columns:
features_list.append(crv_feature3)
else:
pass
if crv_feature4 in well_train.columns:
features_list.append(crv_feature4)
else:
pass
if crv_feature5 in well_train.columns:
features_list.append(crv_feature5)
else:
pass
if crv_feature6 in well_train.columns:
features_list.append(crv_feature6)
else:
pass
if crv_feature7 in well_train.columns:
features_list.append(crv_feature7)
else:
pass
if crv_feature8 in well_train.columns:
features_list.append(crv_feature8)
else:
pass
if crv_feature9 in well_train.columns:
features_list.append(crv_feature9)
else:
pass
if crv_feature10 in well_train.columns:
features_list.append(crv_feature10)
else:
pass
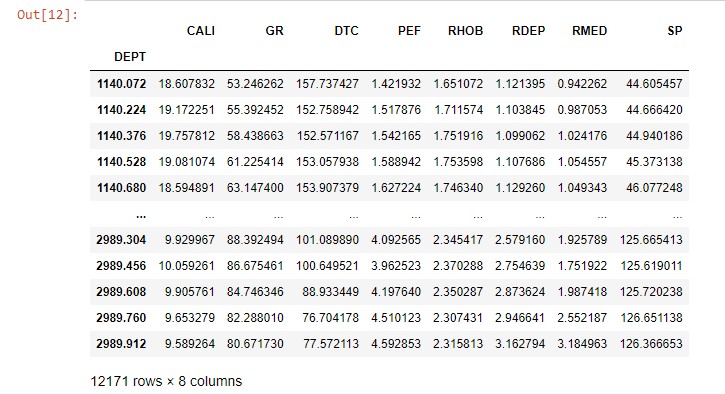
well_train_features = well_train[features_list]
well_train_features
Creación de funciones para graficar el modelo
En la siguiente celda crearemos 2 funciones para graficar los resutados del entrenamiento del modelo. Cada una de ellas creará 2 gráficos, uno donde podremos ver la pérdida o loss, y el otro donde veremos la métrica.
Las 2 funciones son las siguientes:
- get_plot_no_val: En los gráficos veremos los resultados del fit del modelo.
- get_plot_val: En los gráficos veremos los resultados del fit del modelo y de la validación del mismo.
def get_plot_no_val(historia):
plt.rcParams["figure.figsize"] = (14,3.5)
plt.subplots_adjust(wspace=0.5)
plt.subplot(1,2,1)
plt.plot(historia.history[f'{list(historia.history.keys())[0]}'])
#plt.plot(historia.history[f'{list(historia.history.keys())[2]}'])
plt.ylabel(f'Pérdida ({list(historia.history.keys())[0]})')
plt.xlabel('Iteración')
# plt.legend(['Entrenamiento','Validación'])
plt.subplot(1,2,2)
plt.plot(historia.history[f'{list(historia.history.keys())[1]}'])
# plt.plot(historia.history[f'{list(historia.history.keys())[3]}'])
plt.ylabel(f'Métrica ({list(historia.history.keys())[1]})')
plt.xlabel('Iteración')
# plt.legend(['Entrenamiento','Validación'])
ax = plt.gca()
ax.yaxis.set_label_position("right")
ax.yaxis.tick_right()
plt.show()
##############################
def get_plot_val(historia):
plt.rcParams["figure.figsize"] = (14,3.5)
plt.subplots_adjust(wspace=0.5)
plt.subplot(1,2,1)
plt.plot(historia.history[f'{list(historia.history.keys())[0]}'])
plt.plot(historia.history[f'{list(historia.history.keys())[2]}'])
plt.ylabel(f'Pérdida ({list(historia.history.keys())[0]})')
plt.xlabel('Iteración')
plt.legend(['Entrenamiento','Validación'])
plt.subplot(1,2,2)
plt.plot(historia.history[f'{list(historia.history.keys())[1]}'])
plt.plot(historia.history[f'{list(historia.history.keys())[3]}'])
plt.ylabel(f'Métrica ({list(historia.history.keys())[1]})')
plt.xlabel('Iteración')
plt.legend(['Entrenamiento','Validación'])
ax = plt.gca()
ax.yaxis.set_label_position("right")
ax.yaxis.tick_right()
plt.show()
Keras: Entrenamiento del modelo (fit)
En esta celda obtendremos nuestro modelo utilizando la librerías scikit-learn y Keras.
Para comenzar, utilizaremos StandarScaler para estandarizar los datos de las features eliminando la media y escalándolos de forma que su varianza sea igual a 1. Acto seguido, dividiremos con train_test_split los datos, en un conjunto que utilizaremos en la creación del modelo (x_train, y_train), y otro que utilizaremos para validar dicho modelo (x_test, y_test). El método fit de la librería Keras será el encargado de entrenar el modelo.
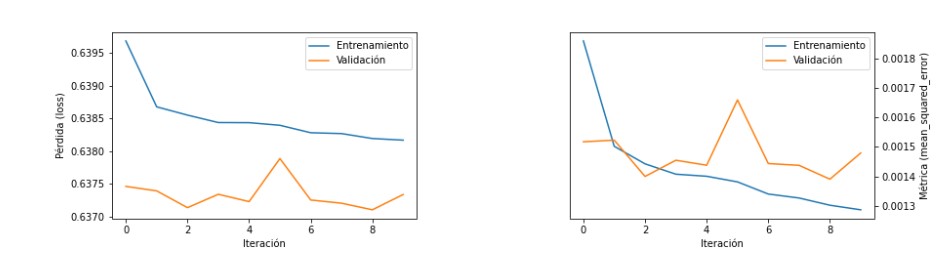
Luego, llamaremos una de las funciones para graficar y plotearemos dos gráficos, uno con la pérdida, y el otro con la métrica.
⚠️ El modelo creado en esta celda es sólo una variable dentro el cuaderno jupyter notebook, o sea, si volvemos a correr la celda, o si eliminamos la variable que contiene el modelo de alguna manera, lo borraremos PERMANENTEMENTE. Para obtener información sobre cómo guardar un modelo creado con Keras para poder utilizarlo en otro momento, por favor visiten: https://keras.io/api/models/model_saving_apis/model_saving_and_loading/ .
ℹ️ Para mayor información sobre StandarScaler y train_test_split visiten la págin web https://scikit-learn.org/stable/index.html .
ℹ️ Para mayor información sobre el método fit por favor visiten la págin web de Keras https://keras.io/api/models/model_training_apis/#fit-method .
X = well_train_features
Y = well_train['target']
scaler = StandardScaler()
scaler.fit(X)
X = scaler.transform(X)
x_train, x_test, y_train, y_test = train_test_split(X, Y, test_size=0.2, random_state=True)
model = Sequential()
#model.add(Dense(1000, input_dim=len(well_train_features.columns), activation='relu'))
model.add(Dense(1000, activation='relu'))
# model.add(Dropout(0.9))
model.add(Dense(250, activation='relu'))
# model.add(Dropout(0.8))
model.add(Dense(1, activation='sigmoid'))
model.compile(loss='binary_crossentropy', optimizer='adam', metrics=['MeanSquaredError'])
# log_dir = "logs/fit/" + datetime.datetime.now().strftime("%Y%m%d-%H%M%S")
# tensorboard_callback = tf.keras.callbacks.TensorBoard(log_dir=log_dir, histogram_freq=1, update_freq='batch')
historia = model.fit(x_train, y_train, batch_size=10, epochs=10, validation_data=(x_test,y_test))
clear_output()
get_plot_val(historia)
Keras: Aplicación del modelo - Obtención de la curva emulada (predict)
Ahora que tenemos nuestro modelo, vamos a utilizarlo para obtener una curva emulada que debería tener valores similares a los de la curva target que elegimos. Esta tarea la haremos utilizando el método predict de la librería Keras.
ℹ️ Para mayor información sobre predict por favor visiten la págin web de Keras https://keras.io/api/models/model_training_apis/#predict-method .
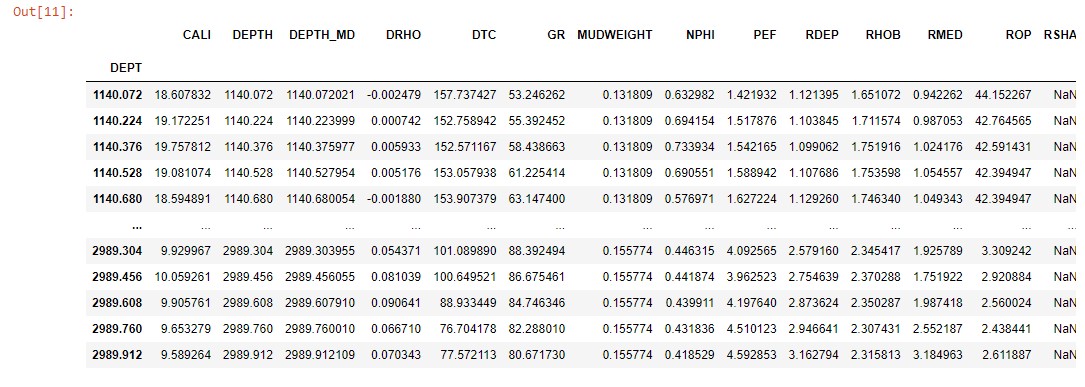
crv_emulated = 'NPHI_emulada'
well_train[crv_emulated]=model.predict(
X,
batch_size=10,
verbose=0,
steps=None,
callbacks=None,
max_queue_size=10,
workers=1,
use_multiprocessing=False)
well_train
Plot para comparar curva target vs. curva emulada
Para finalizar, graficaremos un plot similar al combinado, pero que contenga en el último track, las curvas target y emulated.
top_plot= well_top
bot_plot= well_bot
crv_target = 'NPHI'
crv_emulated = 'NPHI_emulada'
# Escalas curvas
target_izq = .6
target_der = 0
# numero de lineas verticales del track que va a depender de la escala de la curva
ax1_ticks = 6 # Convencionales
ax2_ticks = 6 # Resistividades
ax3_ticks = 7 # Porosidades
ax4_ticks = 7 # Target & Emulated
##################### CODIGO ###################################################
fig, ax = plt.subplots(figsize=(20,20))#Set up the plot axes
ax1 = plt.subplot2grid((1,6), (0,0), rowspan=1, colspan = 1)
ax11 = ax1.twiny()
ax12 = ax1.twiny()
ax2 = plt.subplot2grid((1,6), (0,1), rowspan=1, colspan = 1, sharey = ax1)
ax21 = ax2.twiny()
ax3 = plt.subplot2grid((1,6), (0,2), rowspan=1, colspan = 1, sharey = ax1) #
ax31 = ax3.twiny()
ax32 = ax3.twiny()
ax4 = plt.subplot2grid((1,6), (0,3), rowspan=1, colspan = 1, sharey = ax1) #
ax41 = ax4.twiny()
################################################################################
### CAL ###
cal_izq = 10 # min=izq
cal_der = 20 # max=der
ax1.plot(well_train[crv_cal], well_train.index, color = "black", linewidth = 0.5)
ax1.set_ylabel("Depth (m)")
if abs(top_plot-bot_plot) > 500:
ax1.yaxis.set_major_locator(MaxNLocator(round(abs(top_plot-bot_plot)/99)))
elif abs(top_plot-bot_plot) <= 500 and abs(top_plot-bot_plot) > 200:
ax1.yaxis.set_major_locator(MaxNLocator(round(abs(top_plot-bot_plot)/49)))
else:
ax1.yaxis.set_major_locator(MaxNLocator(round(abs(top_plot-bot_plot)/24)))
ax1.set_xlabel(f"Caliper {well_train[crv_cal].name}")
ax1.set_xlim(cal_izq, cal_der)
ax1.xaxis.label.set_color("black")
ax1.tick_params(axis='x', colors="black")
ax1.spines["top"].set_edgecolor("black")
ax1.set_xticks(np.arange(cal_izq, cal_der+1, ((cal_der-cal_izq)/(ax1_ticks-1))))
ax1.xaxis.set_major_formatter(StrMethodFormatter('{x:.0f}'))
ax1.fill_betweenx(well_train.index, well_train[crv_bit], well_train[crv_cal],
where=well_train[crv_bit]>=well_train[crv_cal], interpolate=True, color='brown')
ax1.fill_betweenx(well_train.index, well_train[crv_bit], well_train[crv_cal],
where=well_train[crv_bit]<=well_train[crv_cal], interpolate=True, color='#a6ecfa')
################################################################################
### GR ###
gr_izq=0
gr_der=200
ax11.plot(well_train[crv_gr], well_train.index, color = "green", linewidth = 1.5)
ax11.set_xlabel(f"Gamma Ray ({well_train[crv_gr].name})")
ax11.xaxis.label.set_color("green")
ax11.set_xlim(gr_izq, gr_der)
ax11.tick_params(axis='x', colors="green")
ax11.spines["top"].set_position(("axes", 1.1))
ax11.spines["top"].set_visible(True)
ax11.spines["top"].set_edgecolor("green")
ax11.title.set_color('green')
ax11.set_xticks(np.arange(gr_izq, gr_der+1, ((gr_der-gr_izq)/(ax1_ticks-1))))
ax11.xaxis.set_major_formatter(StrMethodFormatter('{x:.0f}'))
################################################################################
### SP ###
sp_izq=-80
sp_der=20
ax12.plot(well_train[crv_sp], well_train.index, color = "red", linewidth = 1.5)
ax12.set_xlabel(f"SP ({well_train[crv_sp].name})")
ax12.xaxis.label.set_color("red")
ax12.set_xlim(sp_izq,sp_der)
ax12.tick_params(axis='x', colors="r")
ax12.spines["top"].set_position(("axes", 1.06))
ax12.spines["top"].set_visible(True)
ax12.spines["top"].set_edgecolor("r")
ax12.title.set_color('r')
ax12.set_xticks(np.arange(sp_izq, sp_der+1, ((sp_der-sp_izq)/(ax1_ticks-1))))
ax12.xaxis.set_major_formatter(StrMethodFormatter('{x:.0f}'))
################################################################################
### RESD ###
res_izq=0.1
res_der=100
ax2.plot(well_train[crv_dp_res], well_train.index, color = "blue", linewidth = 2)
ax2.set_xlabel(f"Deep Resistivity ({well_train[crv_dp_res].name})")
ax2.set_xlim(res_izq, res_der)
ax2.xaxis.label.set_color("blue")
ax2.tick_params(axis='x', colors="blue")
ax2.spines["top"].set_edgecolor("blue")
ax2.set_xticks(np.arange(res_izq, res_der+.1, (res_der/(ax2_ticks-1))))
ax2.set_xscale("log")
ax2.grid(True, which="both", axis='x')
ax2.xaxis.set_major_formatter(StrMethodFormatter('{x:.1f}'))
################################################################################
### RESS ###
ax21.plot(well_train[crv_sh_res], well_train.index, color = "r", linewidth = 0.5)
ax21.set_xlabel(f"Shallow Resistivity ({well_train[crv_sh_res].name})")
ax21.set_xlim(res_izq, res_der)
ax21.xaxis.label.set_color("r")
ax21.spines["top"].set_position(("axes", 1.06))
ax21.spines["top"].set_visible(True)
ax21.tick_params(axis='x', colors="r")
ax21.spines["top"].set_edgecolor("r")
ax21.set_xticks(np.arange(res_izq, res_der+.1, ((res_der-res_izq)/(ax2_ticks-1))))
ax21.set_xscale("log")
ax21.xaxis.set_major_formatter(StrMethodFormatter('{x:.1f}'))
################################################################################
### DEN BULK ###
denbulk_izq = 1.65
denbulk_der = 2.65
ax3.plot(well_train[crv_den_bulk], well_train.index, color = "red", linewidth = 1.0)
ax3.set_xlabel(f"Density Bulk ({well_train[crv_den_bulk].name})")
ax3.set_xlim(denbulk_izq,denbulk_der)
ax3.xaxis.label.set_color("red")
ax3.tick_params(axis='x', colors="r")
ax3.spines["top"].set_edgecolor("r")
ax3.set_xticks(np.arange(denbulk_der, denbulk_izq+.01, ((denbulk_izq-denbulk_der)/(ax3_ticks-1))))
ax3.xaxis.set_major_formatter(StrMethodFormatter('{x:.2f}'))
################################################################################
### NEU POR ###
neupor_izq = .6
neupor_der = 0
ax31.plot(well_train[crv_neu_por], well_train.index, color = "blue", linewidth = 1.0)
ax31.set_xlabel(f"Neutron Porosity ({well_train[crv_neu_por].name})")
ax31.xaxis.label.set_color("b")
ax31.set_xlim(neupor_izq,neupor_der)
ax31.tick_params(axis='x', colors="blue")
ax31.spines["top"].set_position(("axes", 1.06))
ax31.spines["top"].set_visible(True)
ax31.spines["top"].set_edgecolor("blue")
ax31.set_xticks(np.arange(neupor_der, neupor_izq+.01, (abs(neupor_der-neupor_izq)/(ax3_ticks-1))))
ax31.xaxis.set_major_formatter(StrMethodFormatter('{x:.2f}'))
################################################################################
### AUX POR ###
auxpor_izq = .6
auxpor_der = 0
auxpor_desc = 'Empty Porosity'
ax32.plot(well_train[crv_nmr_por], well_train.index, color = "black", linewidth = 1.0)
ax32.set_xlabel(f"{auxpor_desc} ({well_train[crv_nmr_por].name})")
ax32.xaxis.label.set_color("black")
ax32.set_xlim(auxpor_izq,auxpor_der)
ax32.tick_params(axis='x', colors="black")
ax32.spines["top"].set_position(("axes", 1.1))
ax32.spines["top"].set_visible(True)
ax32.spines["top"].set_edgecolor("black")
ax32.set_xticks(np.arange(auxpor_der, auxpor_izq+.01, (abs(auxpor_der-auxpor_izq)/(ax3_ticks-1))))
ax32.xaxis.set_major_formatter(StrMethodFormatter('{x:.2f}'))
################################################################################
### TARGET & EMULATED ###
ax4.plot(well_train[crv_target], well_train.index, color = "black", linewidth = 1.5)
ax4.set_xlabel(f"Target ({well_train[crv_target].name})")
ax4.set_xlim(target_izq,target_der)
ax4.xaxis.label.set_color("black")
ax4.tick_params(axis='x', colors="black")
ax4.spines["top"].set_edgecolor("black")
ax4.set_xticks(np.arange(neupor_der, neupor_izq+.01, (abs(neupor_der-neupor_izq)/(ax4_ticks-1))))
ax4.xaxis.set_major_formatter(StrMethodFormatter('{x:.2f}'))
ax41.plot(well_train[crv_emulated], well_train.index, color = "red", linewidth = 0.5)
ax41.set_xlabel(f"Emulated ({well_train[crv_emulated].name})")
ax41.xaxis.label.set_color("r")
ax41.set_xlim(target_izq,target_der)
ax41.tick_params(axis='x', colors="red")
ax41.spines["top"].set_position(("axes", 1.06))
ax41.spines["top"].set_visible(True)
ax41.spines["top"].set_edgecolor("red")
ax41.set_xticks(np.arange(neupor_der, neupor_izq+.01, (abs(neupor_der-neupor_izq)/(ax4_ticks-1))))
ax41.xaxis.set_major_formatter(StrMethodFormatter('{x:.2f}'))
################################################################################
fig.suptitle("CURVA TARGET vs. CURVA EMULADA"+"\n"+"POZO: "+las.well.well.value,size=26,
x=0.035,y=1,ha="left")
# Common functions for setting up the plot can be extracted into
# a for loop. This saves repeating code.
for ax in [ax1, ax2, ax3, ax4]:
ax.set_ylim(bot_plot, top_plot)
ax.grid(which='major', color='lightgrey', linestyle='-')
ax.xaxis.set_ticks_position("top")
ax.xaxis.set_label_position("top")
ax.spines["top"].set_position(("axes", 1.02))
for ax in [ax2, ax3, ax4]:
plt.setp(ax.get_yticklabels(), visible = False)
fig.subplots_adjust(wspace = 0.2)
mplcursors.cursor(hover=True)
plt.tight_layout()
plt.show()
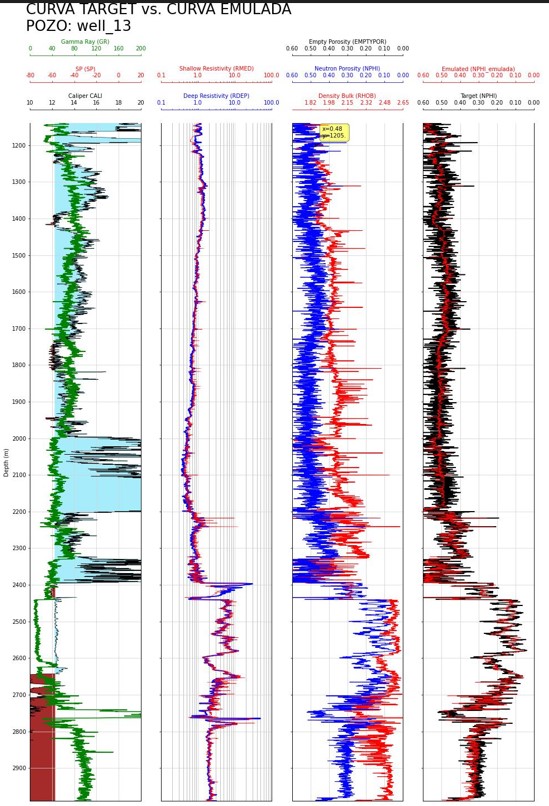
Resumiendo, hemos elegido las curvas para el entrenamiento de la RNA, tanto las features, como la curva target. Luego utilizamos Keras para entrenar, validar, y aplicar el modelo con dichas curvas. Por último, graficamos la curva target vs. la curva emulated para compararlas y poder tomar decisiones sobre si modificamos, o no, el modelo obtenido.
Les agradecemos su tiempo y esperamos fervientemente que hayan disfrutado este artículo. Si tienen alguna consulta, desean hacer algún comentario o sugerencia para mejorar el contenido, o simplemente indicarles qué les pareció este artículo, debajo pueden hacerlo.
Esperamos reencontrarlos en algún otro artículo del sitio. Hasta luego!
Tags
Recent Posts