Emulación de curvas de pozo utilizando RNA - Entrenamiento y Aplicación del modelo con Keras - Parte 2
Uno de los usos más comunes que le dan los petrofísicos y analistas de perfiles a las redes neuronales, según nuestro entender, es la emulación de datos de interés para un pozo de petróleo o gas, a partir del entrenamiento de redes neuronales con datos registrados en ese mismo pozo, y/o en otros pozos cercanos donde las litologías atravesadas en todos ellos sean análogas (mismas formaciones, espesores similares, etc).
Bienvenidas/os!!, muchas gracias por visitarnos, si están comenzando en Python y este es el primer artículo al que entran en nuestro sitio, les recomendamos visitar Primeros Pasos.
ℹ️ Recuerden que todos los ejemplos de códigos en nuestro sitio web van a encontrarlos en nuestro repositorio GitHub. Los códigos de este artículo los encontrarán en ANN_supervised_regression_train-apply.ipynb.
En este artículo seleccionaremos uno de los Pozos de Trabajo definidos utilizando el código python explicado en el artículo Emulación de curvas de pozo utilizando RNA - Entrenamiento y Evaluación del modelo con Keras - Parte 1, para luego transformarlo en Pozo Entrenador. Importaremos sus datos desde el archivo .las con las librerías lasio y pandas. Al dataframe resultante le realizaremos un control de calidad. Para finalizar, visualizaremos sus datos en un plot combinado.
POZO ENTRENADOR
Una vez tengamos la información sobre la cantidad de pozos en los que aparece cada curva, podemos ya elegir convenientemente el Pozo Entrenador.
CARGA Y QC DE LOS DATOS
En los siguientes pasos, importaremos los datos del Pozo Entrenador desde el archivo .las y los depuraremos para que queden listos para el entrenamiento de la red.
INDICE
- Cargamos con lasio el archivo .LAS con los datos del pozo entrenador y creamos un nuevo DF pandas
- Plot QC
- Declaración de variables para plot combinado
- Plot Combinado
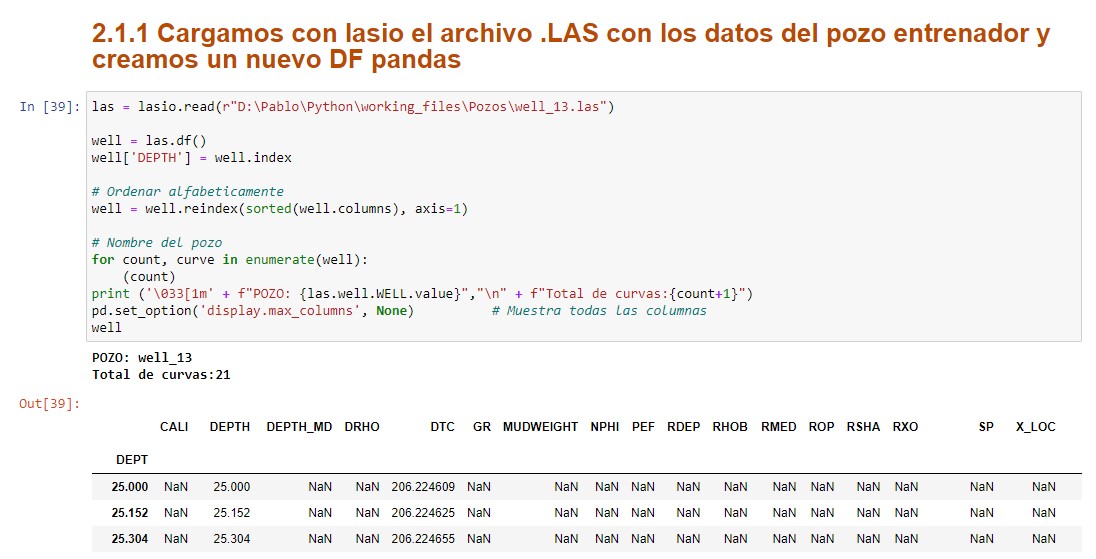
Cargamos con lasio el archivo .LAS con los datos del pozo entrenador y creamos un nuevo DF pandas
Como primera acción, cargaremos los datos del archivo .las a un dataframe. Lo haremos utilizando lasio y pandas. Al dataframe lo llamaremos simplemente well.
En la siguiente celda crearemos el df y le agregaremos una nueva curva llamada DEPTH a partir del index del archivo lasio del cual proviene el df. Además, visualizaremos datos de las columnas, (curvas registradas en el pozo), tal como valores por profundidad, nombre de cada curva. También podremos observar el nombre del pozo y la cantidad de curvas registradas.
ℹ️ Si desean hacer un control de calidad de los datos del dataframe más exhaustivo, pueden chequear métodos de pandas con este fin en el artículo Manejo de datos utilizando pandas.
las = lasio.read(r"D:\Pablo\Python\working_files\Pozos\well_13.las")
well = las.df()
well['DEPTH'] = well.index
# Ordenar alfabeticamente
well = well.reindex(sorted(well.columns), axis=1)
# Nombre del pozo
for count, curve in enumerate(well):
(count)
print ('\033[1m' + f"POZO: {las.well.WELL.value}","\n" + f"Total de curvas:{count+1}")
pd.set_option('display.max_columns', None) # Muestra todas las columnas
well
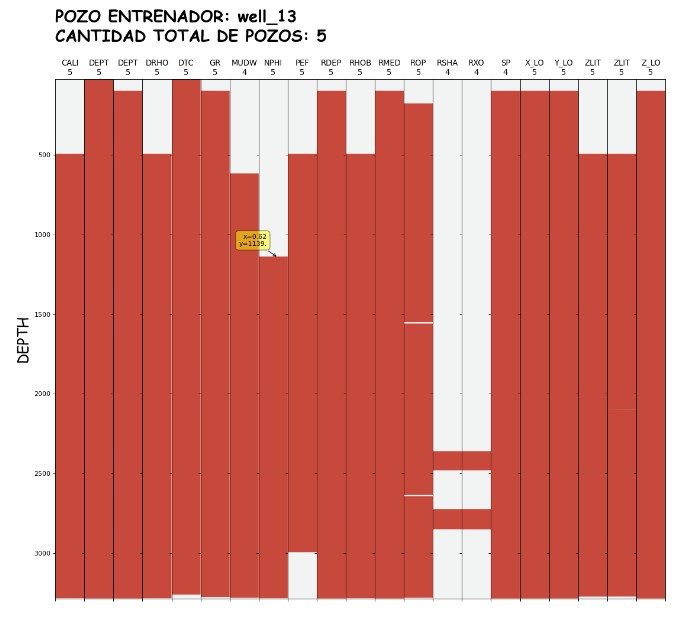
Plot QC
Llegó la hora de graficar los datos del pozo entrenador. Al correr esta celda, vamos a poder visualizar información cualitativa de todas las curvas del pozo entrenador para verificar fácilmente si tienen gaps. Además, si es posible, intentaremos encontrar un tramo que luego podríamos utilizar para el entrenamiento de la RNA.
Según nuestra opinión, el dato más importante presente en este plot, es el número de pozos del conjunto Pozos de trabajo en el cual está presente cada curva. Dicho valor aparecerá debajo del nombre de cada columna/curva en el gráfico.
################## DATOS DE ENTRADA ##################
df = well # Nombre del dataframe pandas que creamos
lasio_file = las # Nombre del archivo lasio que creamos al principio
top_plot_qc = lasio_file.well.STRT.value # Top plot
bot_plot_qc = lasio_file.well.STOP.value # Bottom plot
data_color = '#BD1E0E' # color de relleno de los datos
data_alpha = 0.8 # transparencia del color (0: transparente 1:opaco)
####################### CODIGO #######################
if 'dict_curves' in locals():
pass
else:
dict_curves = {}
df_nan = df.notnull() * 1
fig = plt.subplots(figsize=((len(well.columns)*.8),(len(well.columns)*.7))) #Set up the plot axes
for i in range(len(well.columns)):
ax = plt.subplot2grid((1,len(well.columns)), (0,i), rowspan=1, colspan = 1)
ax.plot(df_nan.iloc[:,i], df_nan.index, lw=0)
ax.set_ylim(bot_plot_qc, top_plot_qc)
ax.set_xlim(0, 1)
if dict_curves.get(df_nan.columns[i]) == None:
ax.set_title(df_nan.columns[i][:4], rotation=0) # titulo columnas: [:4] solo muetras primeras 4 letras
# titulo columnas: rotation=0 horizontal, rotation=90 vertical
else:
ax.set_title(label=f'{df_nan.columns[i][:4]}\n{dict_curves.get(df_nan.columns[i])}', rotation=0)
ax.set_facecolor('#F2F4F4')
ax.fill_betweenx(df_nan.index, 0, df_nan.iloc[:,i], facecolor= data_color, alpha= data_alpha)
plt.setp(ax.get_xticklabels(), visible = False)
if i > 0:
plt.setp(ax.get_yticklabels(), visible = False)
else: # Sets the label for the leftmost subplot
ax.set_ylabel('DEPTH', fontsize=22,family='cursive')
plt.subplots_adjust(wspace=0)
plt.suptitle(t=f'\nPOZO ENTRENADOR: {lasio_file.well.WELL.value}\nCANTIDAD TOTAL DE POZOS: {len(las_list)}',
size=25,ha="left", x=0.125, y=1.01, weight=900,family='cursive')
## The font.weight property has effectively 13 values: normal, bold,
## bolder, lighter, 100, 200, 300, ..., 1000. Normal=400, bold=700
mplcursors.cursor(hover=True)
plt.show()
Declaración de variables para plot combinado
Antes de correr la celda para visualizar el plot combinado, vamos a declarar las variables a plotear. Esto lo hacemos por 2 razones:
- Los nombres de las curvas son diferentes según la compañía de perfilaje.
- En el caso de que no contemos con una de las curvas incluidas en el plot, esta celda va a generarla con valores nulos (esto es porque si matplotlib no encuentra ninguna curva, va a dar error y no va a plotear nada).
ℹ️ Incluimos un par de celdas más en el notebook, las cuales contienen código que nos va a permitir chequear las curvas que creamos con la celda de Declaración de variables por posibles errores y/u omisiones, y además, tratar de solucionarlos. Estas celdas no están incluidas en este artículo, pero sí en ANN_supervised_regression_train-apply.ipynb.
##################################################
# #
# DECLARACION DE VARIABLES PARA PLOT COMBINADO #
# #
##################################################
bitsize = 12.25
## ax1
crv_cal = 'CALI' # CALIPER
crv_bit = 'BIT' # BIT SIZE
crv_gr = 'GR' # GAMMA RAY
crv_sp = 'SP' # SPONTANEOUS POTENTIAL
# ax2
crv_dp_res = 'RDEP' # DEEP RESISTIVITY
crv_sh_res = 'RMED' # SHALLOW RESISTIVITY
#ax3
crv_neu_por = 'NPHI' # NEUTRON POROSITY
crv_den_bulk = 'RHOB' # BULK DENSITY
crv_nmr_por = 'EMPTYPOR' # OTHER POROSITY
#ax4
crv_con = 'DCON' # CONDUCTIVITY
##############################################################
if crv_cal in well.columns:
pass
else:
well[crv_cal]=np.nan
##############
if crv_bit in well.columns:
pass
else:
well[crv_bit]=bitsize
##############
if crv_gr in well.columns:
pass
else:
well[crv_gr]=np.nan
##############
if crv_sp in well.columns:
pass
else:
well[crv_sp]=np.nan
##############
if crv_dp_res in well.columns:
pass
else:
well[crv_dp_res]=np.nan
##############
if crv_sh_res in well.columns:
pass
else:
well[crv_sh_res]=np.nan
##############
if crv_neu_por in well.columns:
pass
else:
well[crv_neu_por]=np.nan
##############
if crv_nmr_por in well.columns:
pass
else:
well[crv_nmr_por]=np.nan
##############
if crv_den_bulk in well.columns:
pass
else:
well[crv_den_bulk]=np.nan
##############
if crv_con in well.columns:
pass
else:
well[crv_con]=1000/well[crv_dp_res]
################
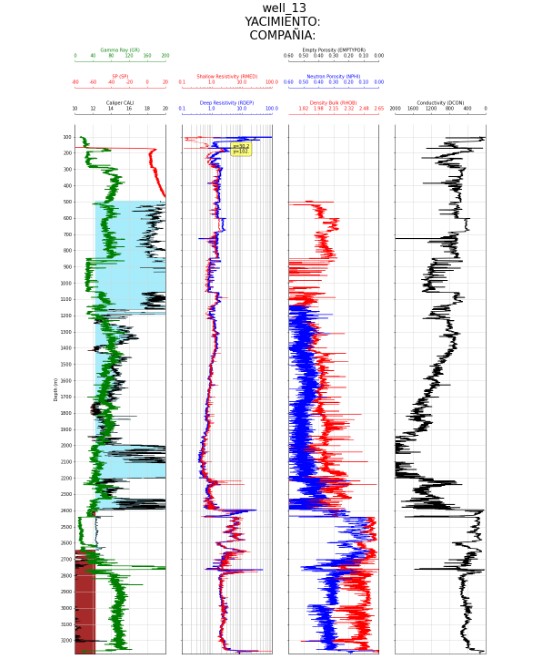
Plot Combinado
En la celda de abajo está el código para plotear 4 tracks:
- Curvas convencionales(GR,CAL,BIT,SP)
- Resistividades(RDEP,RMED)
- Porosidades(BULKDEN,PORNEU,AUXPOR)
- Conductividad (DCON)
Además, deberíamos tener en cuenta lo siguiente:
- Para cambiar TOP y BOT del plot, modificar los valores top_plot y bot_plot.
- La cantidad de labels de cada track se modifica con las variables ax1_ticks,ax2_ticks,etc
- El sentido de las escalas de las curvas depende de cada una de ellas. Por esto, en las escalas no definimos valores mínimo y máximo, sino valores izquierda y derecha. (Por ejemplo los límites para la curva gr son gr_izq=0, gr_der=200, pero para la curva Porosidad Neutron son neupor_izq = .6, neupor_der = 0).
top_plot= las.well.STRT.value
bot_plot= las.well.STOP.value
## numero de lineas verticales del track que va a depender de la escala de la curva
ax1_ticks = 6 # Convencionales
ax2_ticks = 6 # Resistividades
ax3_ticks = 7 # Porosidades
ax4_ticks = 6 # Conductividad
fig, ax = plt.subplots(figsize=(20,20))#Set up the plot axes
ax1 = plt.subplot2grid((1,6), (0,0), rowspan=1, colspan = 1)
ax11 = ax1.twiny()
ax12 = ax1.twiny()
ax2 = plt.subplot2grid((1,6), (0,1), rowspan=1, colspan = 1, sharey = ax1)
ax21 = ax2.twiny()
ax3 = plt.subplot2grid((1,6), (0,2), rowspan=1, colspan = 1, sharey = ax1) #
ax31 = ax3.twiny()
ax32 = ax3.twiny()
ax4 = plt.subplot2grid((1,6), (0,3), rowspan=1, colspan = 1, sharey = ax1) #
################################################################################
### CAL ###
cal_izq = 10 # min=izq
cal_der = 20 # max=der
ax1.plot(well[crv_cal], well.index, color = "black", linewidth = 0.5)
ax1.set_ylabel("Depth (m)")
if abs(top_plot-bot_plot) > 500:
ax1.yaxis.set_major_locator(MaxNLocator(round(abs(top_plot-bot_plot)/99)))
elif abs(top_plot-bot_plot) <= 500 and abs(top_plot-bot_plot) > 200:
ax1.yaxis.set_major_locator(MaxNLocator(round(abs(top_plot-bot_plot)/49)))
else:
ax1.yaxis.set_major_locator(MaxNLocator(round(abs(top_plot-bot_plot)/24)))
ax1.set_xlabel(f"Caliper {well[crv_cal].name}")
ax1.set_xlim(cal_izq, cal_der)
ax1.xaxis.label.set_color("black")
ax1.tick_params(axis='x', colors="black")
ax1.spines["top"].set_edgecolor("black")
ax1.set_xticks(np.arange(cal_izq, cal_der+1, ((cal_der-cal_izq)/(ax1_ticks-1))))
ax1.xaxis.set_major_formatter(StrMethodFormatter('{x:.0f}'))
ax1.fill_betweenx(well.index, well[crv_bit], well[crv_cal],
where=well[crv_bit]>=well[crv_cal], interpolate=True, color='brown')
ax1.fill_betweenx(well.index, well[crv_bit], well[crv_cal],
where=well[crv_bit]<=well[crv_cal], interpolate=True, color='#a6ecfa')
################################################################################
### GR ###
gr_izq=0
gr_der=200
ax11.plot(well[crv_gr], well.index, color = "green", linewidth = 1.5)
ax11.set_xlabel(f"Gamma Ray ({well[crv_gr].name})")
ax11.xaxis.label.set_color("green")
ax11.set_xlim(gr_izq, gr_der)
ax11.tick_params(axis='x', colors="green")
ax11.spines["top"].set_position(("axes", 1.12))
ax11.spines["top"].set_visible(True)
ax11.spines["top"].set_edgecolor("green")
ax11.title.set_color('green')
ax11.set_xticks(np.arange(gr_izq, gr_der+1, ((gr_der-gr_izq)/(ax1_ticks-1))))
ax11.xaxis.set_major_formatter(StrMethodFormatter('{x:.0f}'))
################################################################################
### SP ###
sp_izq=-80
sp_der=20
ax12.plot(well[crv_sp], well.index, color = "red", linewidth = 1.5)
ax12.set_xlabel(f"SP ({well[crv_sp].name})")
ax12.xaxis.label.set_color("red")
ax12.set_xlim(sp_izq,sp_der)
ax12.tick_params(axis='x', colors="r")
ax12.spines["top"].set_position(("axes", 1.07))
ax12.spines["top"].set_visible(True)
ax12.spines["top"].set_edgecolor("r")
ax12.title.set_color('r')
ax12.set_xticks(np.arange(sp_izq, sp_der+1, ((sp_der-sp_izq)/(ax1_ticks-1))))
ax12.xaxis.set_major_formatter(StrMethodFormatter('{x:.0f}'))
################################################################################
### RESD ###
res_izq=0.1
res_der=100
ax2.plot(well[crv_dp_res], well.index, color = "blue", linewidth = 2)
ax2.set_xlabel(f"Deep Resistivity ({well[crv_dp_res].name})")
ax2.set_xlim(res_izq, res_der)
ax2.xaxis.label.set_color("blue")
ax2.tick_params(axis='x', colors="blue")
ax2.spines["top"].set_edgecolor("blue")
ax2.set_xticks(np.arange(res_izq, res_der+.1, (res_der/(ax2_ticks-1))))
ax2.set_xscale("log")
ax2.grid(True, which="both", axis='x')
ax2.xaxis.set_major_formatter(StrMethodFormatter('{x:.1f}'))
################################################################################
### RESS ###
ax21.plot(well[crv_sh_res], well.index, color = "r", linewidth = 0.5)
ax21.set_xlabel(f"Shallow Resistivity ({well[crv_sh_res].name})")
ax21.set_xlim(res_izq, res_der)
ax21.xaxis.label.set_color("r")
ax21.spines["top"].set_position(("axes", 1.07))
ax21.spines["top"].set_visible(True)
ax21.tick_params(axis='x', colors="r")
ax21.spines["top"].set_edgecolor("r")
ax21.set_xticks(np.arange(res_izq, res_der+.1, ((res_der-res_izq)/(ax2_ticks-1))))
ax21.set_xscale("log")
ax21.xaxis.set_major_formatter(StrMethodFormatter('{x:.1f}'))
################################################################################
### DEN BULK ###
denbulk_izq = 1.65
denbulk_der = 2.65
ax3.plot(well[crv_den_bulk], well.index, color = "red", linewidth = 1.0)
ax3.set_xlabel(f"Density Bulk ({well[crv_den_bulk].name})")
ax3.set_xlim(denbulk_izq,denbulk_der)
ax3.xaxis.label.set_color("red")
ax3.tick_params(axis='x', colors="r")
ax3.spines["top"].set_edgecolor("r")
ax3.set_xticks(np.arange(denbulk_der, denbulk_izq+.01, ((denbulk_izq-denbulk_der)/(ax3_ticks-1))))
ax3.xaxis.set_major_formatter(StrMethodFormatter('{x:.2f}'))
################################################################################
### NEU POR ###
neupor_izq = .6
neupor_der = 0
ax31.plot(well[crv_neu_por], well.index, color = "blue", linewidth = 1.0)
ax31.set_xlabel(f"Neutron Porosity ({well[crv_neu_por].name})")
ax31.xaxis.label.set_color("b")
ax31.set_xlim(neupor_izq,neupor_der)
ax31.tick_params(axis='x', colors="blue")
ax31.spines["top"].set_position(("axes", 1.07))
ax31.spines["top"].set_visible(True)
ax31.spines["top"].set_edgecolor("blue")
ax31.set_xticks(np.arange(neupor_der, neupor_izq+.01, (abs(neupor_der-neupor_izq)/(ax3_ticks-1))))
ax31.xaxis.set_major_formatter(StrMethodFormatter('{x:.2f}'))
################################################################################
### AUX POR ###
auxpor_izq = .6
auxpor_der = 0
auxpor_desc = 'Empty Porosity'
ax32.plot(well[crv_nmr_por], well.index, color = "black", linewidth = 1.0)
ax32.set_xlabel(f"{auxpor_desc} ({well[crv_nmr_por].name})")
ax32.xaxis.label.set_color("black")
ax32.set_xlim(auxpor_izq,auxpor_der)
ax32.tick_params(axis='x', colors="black")
ax32.spines["top"].set_position(("axes", 1.12))
ax32.spines["top"].set_visible(True)
ax32.spines["top"].set_edgecolor("black")
ax32.set_xticks(np.arange(auxpor_der, auxpor_izq+.01, (abs(auxpor_der-auxpor_izq)/(ax3_ticks-1))))
ax32.xaxis.set_major_formatter(StrMethodFormatter('{x:.2f}'))
################################################################################
### CONDUCTIVITY ###
cond_izq = 2000
cond_der = 0
ax4.plot(well[crv_con], well.index, color = "black", linewidth = 1.5)
ax4.set_xlabel(f"Conductivity ({well[crv_con].name})")
ax4.set_xlim(cond_izq,cond_der)
ax4.xaxis.label.set_color("black")
ax4.tick_params(axis='x', colors="black")
ax4.spines["top"].set_edgecolor("black")
ax4.set_xticks(np.arange(cond_der, cond_izq+1, (abs(cond_der-cond_izq)/(ax4_ticks-1))))
ax4.xaxis.set_major_formatter(StrMethodFormatter('{x:.0f}'))
################################################################################
fig.suptitle(las.well.WELL.value+"\n"+"YACIMIENTO: "+las.well.FLD.value+"\n"+"COMPAÑIA: "+las.well.COMP.value,size=26,
x=0.36,y=1)
# Common functions for setting up the plot can be extracted into
# a for loop. This saves repeating code.
for ax in [ax1, ax2, ax3, ax4]:
ax.set_ylim(bot_plot, top_plot)
ax.grid(which='major', color='lightgrey', linestyle='-')
ax.xaxis.set_ticks_position("top")
ax.xaxis.set_label_position("top")
ax.spines["top"].set_position(("axes", 1.02))
for ax in [ax2, ax3, ax4]:
plt.setp(ax.get_yticklabels(), visible = False)
fig.subplots_adjust(wspace = 0.2)
mplcursors.cursor(hover=True)
plt.tight_layout()
plt.show()
En resumen, creamos un dataframe pandas importando los datos de un archivo .las que elegimos como Pozo Entrenador. A continuación, le realizamos un QC y lo visualizamos con un par de gráficos para tratar de determinar cuáles curvas podrían sernos útiles más adelante para el entrenamiento de la RNA. El código python necesario para realizar dicho entrenamiento, lo detallaremos en el artículo Emulación de curvas de pozo utilizando RNA - Entrenamiento y Evaluación del modelo con Keras - Parte 3.
Les agradecemos su tiempo y esperamos fervientemente que hayan disfrutado este artículo. Si tienen alguna consulta, desean hacer algún comentario o sugerencia para mejorar el contenido, o simplemente indicarles qué les pareció este artículo, debajo pueden hacerlo.
Esperamos reencontrarlos en algún otro artículo del sitio. Hasta luego!
Tags
Recent Posts